撰稿:陆亚娴 审校:杨晓燕
休眠微生物广泛存在于各种生境中,这些微生物重要的生态功能不可忽略,但目前还没有比较好的方法识别它们。对休眠微生物识别的不充分严重影响了微生物多样性监测,甚至影响了我们对微生物多样性格局的认识。
东喜院微生态研究团队通过对泡菜微生物体系的研究,提出了一种新的休眠微生物识别方法- Revived Amplicon Sequence Variants Monitoring。报道该方法的研究论文题为“Revived Amplicon Sequence Variants Monitoring in Closed Systems Identifies More Dormant Microorganisms”,于2023年3月15日在《microorganisms》上发表。

图1 文章首页信息
我们的研究团队构建了封闭的泡菜微宇宙体系,排除了扩散等因素的影响,对其中的微生物开展了为期60天的监测。通过对泡菜样品重复多次样品并测序,筛选出丰度在有-无值波动的ASV,并将其识别为休眠微生物(图2)。同时我们将新的识别方法(RAM)与目前常被使用的基因功能预测(GFP)进行了比较,结果显示,我们提出的RAM能够识别比GFP更多的休眠微生物(图3)。
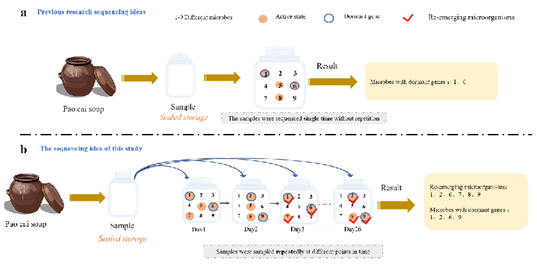
图2 休眠微生物识别方法
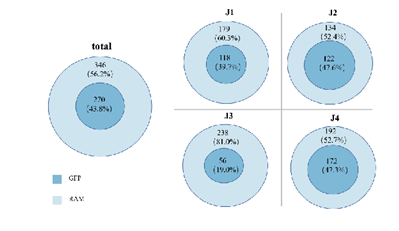
图3 不同时间阶段下GFP和RAM识别到的细菌分类属数量变化
此外,我们的结果还发现RAM和GFP识别到的休眠微生物存在一致的阶段性变化趋势,这表明用RAM识别休眠微生物是可行且高效的。
值得注意的是,RAM和GFP两种休眠微生物识别方法是相互补充和相互促进的,RAM识别到的休眠微生物进行休眠基因水平的研究,以提高GFP对休眠微生物的识别率,从而降低成本。如果结合这两种方法,那么未来对休眠微生物的识别及其生态功能和意义的研究将会得到更好的发展。
论文作者:
陆亚娴(大理大学东喜玛拉雅研究院2022级研究生)
邓巍(大理大学东喜玛拉雅研究院2020级研究生)
祁福良(大理大学东喜玛拉雅研究院2021级本科生)
杨晓燕(大理大学东喜玛拉雅研究院教授)
肖文(大理大学东喜玛拉雅研究院研究员)
论文链接:

